作者:苏打水
导读:
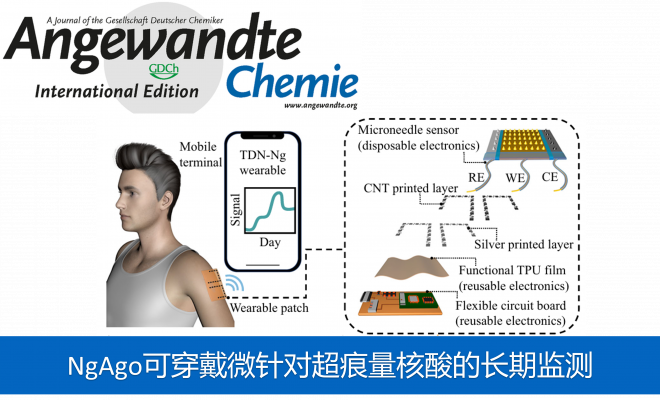
近日,复旦大学方雪恩教授团队在Nat. Commun.中发表论文,报道了一种基于四面体纳米结构的嗜盐菌Argonaute(NgAgo)构建的可穿戴微针传感器,用于长期稳定监测体内败血症的超痕量未扩增核酸。
Long-Term Monitoring of Ultratrace Nucleic Acids Using Tetrahedral Nanostructure-Based Ngago on Wearable Microneedles.
Yang, B.; Wang, H.; Kong, J.; Fang, X. Nat. Commun. 2024, 15 (1), 1936. DOI: 10.1038/s41467-024-46215-w
正文:
近年来,基因检测技术的发展为核酸诊断识别疾病提供了强大工具。其中PCR、等温核酸扩增和CRISPR技术结合的全基因组测序可提供多种病理生理信息,但大多技术均面临周转时间长、设备笨重等问题[1]。新兴的可穿戴技术在个性化医疗、医疗物联网和实时疾病诊断中提供了变革性技术,可能解决传统核酸分子诊断局限。这类先进的可穿戴设备可以监测深层组织、心脏功能、慢性感染性伤口、体温调节和肿瘤消退,以及血糖、水分流失和汗液电解质监测[2]。可穿戴设备可能为持续监测提供新途径,但在ICU场景中仍存在局限,特别是在监测低丰度疾病相关游离DNA或RNA生物标志物方面。
基于此,复旦大学方雪恩教授团队报告了基于四面体 DNA 纳米结构 (TDN) 和原核 argonaute 技术的完全集成的可穿戴电子设备,实时监测由 EBV、金黄色葡萄球菌和铜绿假单胞菌 (staphylococcus aureus, SA) and pseudomonas aeruginosa, PA)引起的脓毒症相关的cfDNA和RNA,从而扩大了在ICU情况下发病时监测脓毒症的可能性。
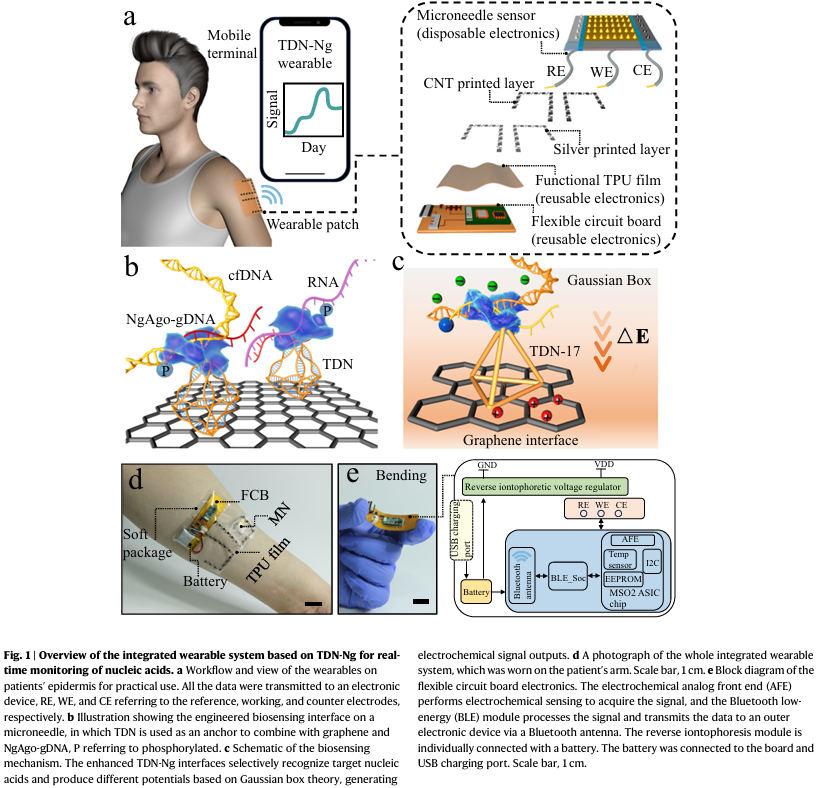
Fig.1a-e详细介绍了该集成式可穿戴设备的各部件组成和传感原理。该可穿戴系统由一次性微针贴片和可重复使用的电子设备(无线柔性电路板、功能性热塑性聚氨酯薄膜)组成。其中,一次性微针(Microneedle, MN )贴片由三电极阵列以紧凑的方式制三合一贴片。采用喷印和铸造技术制备了由第一层银和第二层碳纳米管组成的功能性热塑性聚氨酯(Thermoplastic polyurethane, TPU)。将具有阳极和阴极图案的功能化TPU膜与无线柔性电路板(flexible circuit board, FCB)单独连接,使其能够通过反向离子电泳富集核酸。集成可穿戴系统的第三部分是自行设计和定制的FCB,用于电源和信号管理。
TDN-17被用作固定在石墨烯表面的最佳纳米支架,在TDN-17上,NgAgo蛋白作为一种原核蛋白被固定形成NgAgo/引导DNA复合物,记作NgAgo- gDNA。在gDNA (24nt 5 ‘ -磷酸化单链DNA)的指导下,通过碱基配对搜索整个目标序列。一旦匹配,固定化的NgAgo-gDNA就可以与目标核酸结合,由于候选gDNA的存在,这种结合具有明确的稳定性。
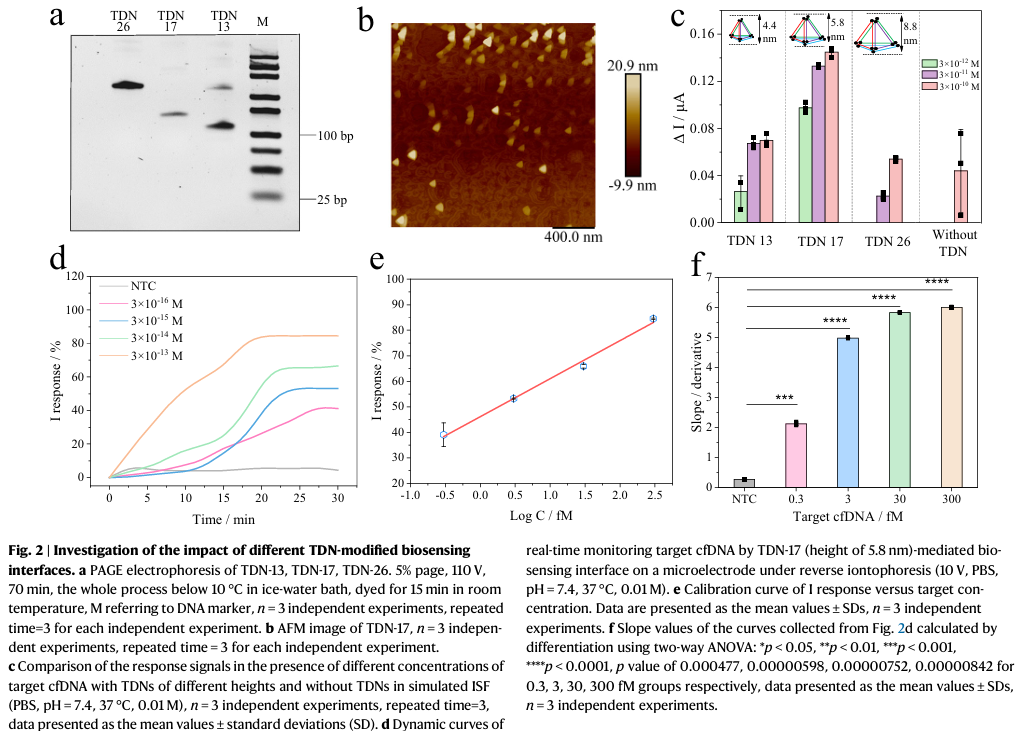
为优化TDN修饰的生物传感界面,作者制备了修饰有不同高度TDNs的生物传感界面,并在TDNs顶端锚定NgAgo-gDNA复合物。如图2c,与没有TDN的系统相比,具有TDN的生物传感系统在~0.28V的电位下电流信号增强,且信号强度随靶标浓度的增加而提高。结果表明,使用高度为5.8 nm的TDN-17时产生的电流信号最强,且检测限与不含TDN的对照组相比降低了两个数量级,达到3×10-12 M。当TDN高度继续增加时,输出信号的强度和检测灵敏度均降低。这证明在合适的高度范围内,空间高度对界面的Donnan电位有一定的调节作用。图 2d-f 使用商用微电极对芯片上TDN-17/NgAgo-gDNA 的检测限进行研究(10 V反向离子电渗),结果表明芯片上TDN-17/NgAgo-gDNA检测限可达3×10-16 M。
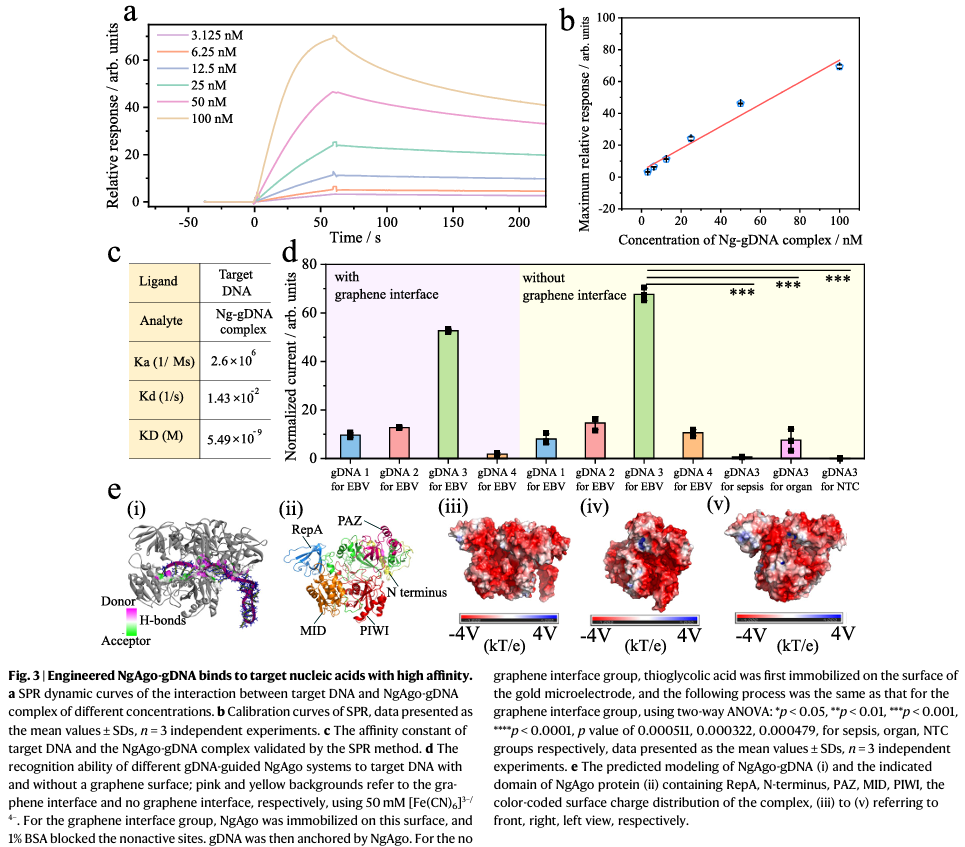
工程DNA引导的NgAgo系统能否识别和结合靶核酸,这对后续的研究具有重要意义。基pAgo技术的原理,推测复合物可能在gDNA的引导下搜索整个核酸序列,其中一个5 ‘ -磷酸化的24-nt特异性序列与目标匹配。一旦匹配,复合体就可以直接与目标特异性结合。作者通过表面等离子体共振(SPR)测试了NgAgo-gDNA与靶标核酸结合亲和力(Fig.3a-c)。结果显示,EBV cfDNA靶标浓度在3.125 nM-100 nM内时,NgAgo-gDNA 复合物的相对响应强度与靶标浓度成线性关系。NgAgo-gDNA 复合物对EBV cfDNA的KD为5.49×10-9 M-1,该系统具有高靶向亲和力,且略强于 CRISPR-dCas9(KD为1×10-9 M-1)[3]。为了消除石墨烯表面的干扰,作者构建了一个简单的电化学生物传感器来进一步研究其结合亲和力。在Fig.3d中,与NTC组相比,gDNA 3组有明显的信号响应,且差异显著。此外,无论石墨烯界面如何,gDNA 3组都具有最大电流。Fig.3e为NgAgo-gDNA的预测模型。NgAgo蛋白具有典型的N末端、PAZ、MID、PIWI结构域以及RepA结构域。gDNA与由N末端、PAZ、MID和PIWI形成的口袋稳定结合。NgAgo-gDNA复合物表面大部分带有负电荷,而RepA结构域的表面则带有正电荷,使其适合于与带负电荷的底物(如DNA或RNA)相互作用。以上结果证明Ng系统能够有效地识别并结合EBV cfDNA靶标。
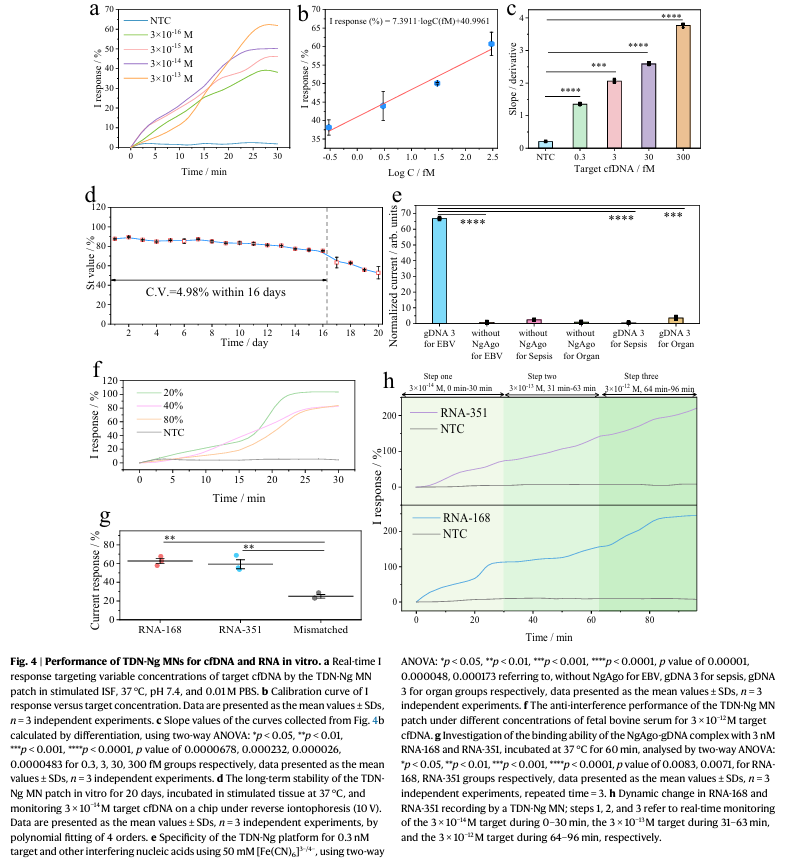
随后,作者验证了TDN-Ng MNs在体外实验中的灵敏度、定量检测、长期稳定性、特异性和抗干扰性能(Fig.4a-f)。TDN-Ng MNs可持续识别并结合靶标 cfDNA,检测限为 3×10-16 M。含靶标cfDNA的实验组和NTC之间存在显著差异,且电流响应对时间的关系曲线的斜率与靶标cfDNA 浓度成正比。此外,结果表明反应在30 min内逐渐趋于稳定,比该团队之前报道的CRISPR MN贴片的监测时间(75 min)更短,表明该研究所构建的基于TDN的NgAgo-gDNA系统的反应动力学更快。为验证TDN-Ng MNs检测靶标核酸的长期稳定性,作者将TDN-Ng MN贴片保存在间质液(ISF,37°C,pH 7.4)中,并在10V反向离子电渗条件下监测3×10−14M的靶标核酸。在前16天内,St值保持稳定,C.V.为4.98%。虽然从第17天开始St值下降,但仍可观察到明显的“S”形信号曲线。以上结果表明,TDN-Ng MN贴片用于体外核酸监测时在合理的C.V.范围内表现出16天的长期稳定性。此外,结果还证明 TDN-Ng MN贴片可以耐受0.3 nM的非特异性核酸和80%的胎牛血清(FBS)干扰,特异性和抗干扰性能好。与人体ISF的总蛋白浓度(20.6 mg/mL)相比,这种对80%FBS(23 mg/mL)的优异抗干扰能力可能使TDN Ng-MN贴片应用于实际。
根据先前的研究,NgAgo-gDNA复合物被报道具有敲低基因和诱导转录沉默的能力。因此,作者怀疑RNA也可能被他们的TDN-Ng系统识别。作者首先筛选RNA-168和RNA-351序列作为疾病相关生物标志物,然后使用工程化的NgAgo-gDNA在ISF中鉴定这两种RNA。在Fig.4g,h中,与对照组相比,RNA-168和RNA-351与NgAgo-gDNA复合物结合并诱导了显著的电流反应。在本研究中,作者选择RNA-168和RNA-351作为靶标,验证TDN-Ng MN贴片的实时监测能力。在3×10−14 M靶标的第一步,随着RNA-168或RNA-351浓度的增加,信号增加。信号在第2步(3×10−13 M)和第3步(3×10−12 M)连续增加,表明该系统能够实时监测靶RNA的动态变化。TDN-Ng MN平台还可以实时监测其他应用,如金黄色葡萄球菌和铜绿假单胞菌。
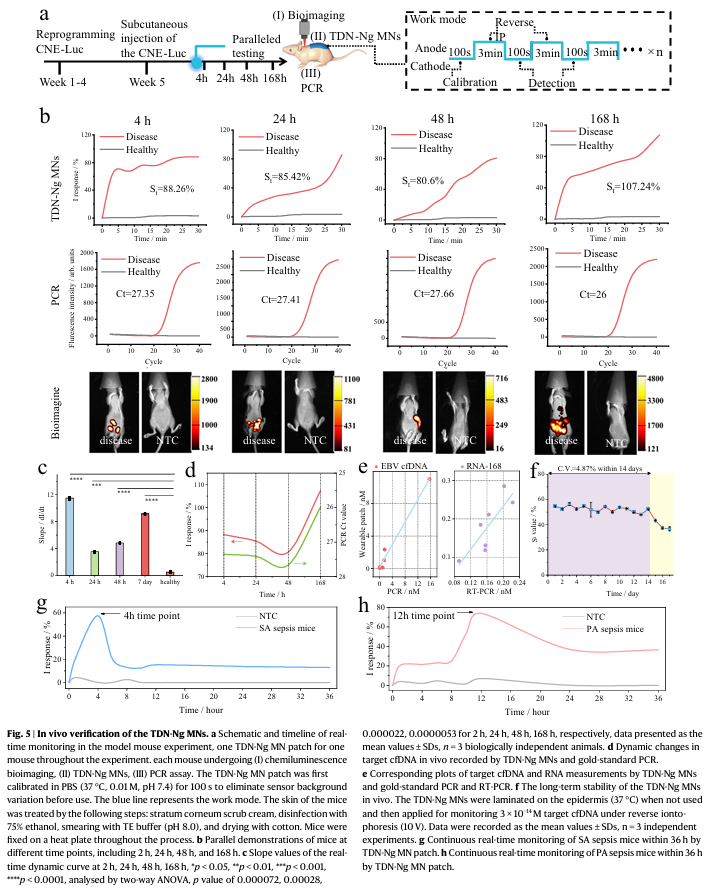
最后,作者进一步验证了TDN-Ng MNs对体内核酸的实时监测性能。据报道,EBV 相关核酸生物标志物与脓毒症有关,且CNE细胞系中存在cfDNA或RNA。因此,作者首先用荧光素酶报告基因(Luc)对CNE细胞系进行重编程,然后将其皮下接种到8周大的雌性BALB/c裸鼠体内以构建EBV-小鼠模型。接着,作者将TDN-Ng MN贴片用于上述小鼠模型。结果如Fig.5a-h显示,TDN-Ng MN贴片与金标准RT-PCR监测结果一致,TDN-Ng MN贴片可用于EBV模型小鼠体内RNA靶标的实时监测,且用于体内核酸实时监测的TDN-Ng MN贴片的在16天内具有可靠稳定性,可满足ICU患者疾病监测的临床要求。
总结:
综上所述,本研究提出了无线集成可穿戴电子设备,通过工程生物传感接口实时监测败血症相关动物模型的纵向核酸。基于TDN和Ng系统的协同作用,可穿戴系统能够连续跟踪体内无细胞DNA和RNA靶点的动态变化,灵敏度为0.3 fM,在体内16天内具有可靠的稳定性。作者还讨论了TDN-17随空间高度增加对TDN-Ng系统的灵敏度增强和实时监测动态平衡的机理。目前这项工作的无扩增策略代表了基于核酸的可穿戴设备的飞跃,并加速了用于ICU医疗保健管理的下一代生物传感器的出现。这项工作的后续发展将集中在直接记录波动的输入信号(增加,减少和恢复),更高亲和力的构建,生物传感膜再生和自动数据采集。同时,基于人工智能的机器学习可用于协助预测ICU医疗报警管理的数据采集。
参考文献:
- [1] P. Fozouni, S. Son, M. Díaz de León Derby, G. J. Knott, C. N. Gray, M. V. D’Ambrosio, C. Zhao, N. A. Switz, G. R. Kumar, S. I. Stephens, D. Boehm, C.-L. Tsou, J. Shu, A. Bhuiya, M. Armstrong, A. R. Harris, P.-Y. Chen, J. M. Osterloh, A. Meyer-Franke, B. Joehnk, K. Walcott, A. Sil, C. Langelier, K. S. Pollard, E. D. Crawford, A. S. Puschnik, M. Phelps, A. Kistler, J. L. DeRisi, J. A. Doudna, D. A. Fletcher, M. Ott, Cell 2021, 184, 323-333.e329. DOI: 10.1016/j.cell.2020.12.001
- [2] D. S. Yang, R. Ghaffari, J. A. Rogers, Science 2023, 379, 760-761. DOI: 10.1126/science.abq5916
- [3] J. Yang, Y. Song, X. Deng, J. A. Vanegas, Z. You, Y. Zhang, Z. Weng, L. Avery, K. D. Dieckhaus, A. Peddi, Y. Gao, Y. Zhang, X. Gao, Nat. Chem. Biol. 2023, 19, 45-54. DOI:10.1038/s41589-022-01135-y
本文版权属于 Chem-Station化学空间, 欢迎点击按钮分享,未经许可,谢绝转载.



















No comments yet.